Emmy und Meggie, zwei FAU-Supercomputer
Im Land der Rechenriesen
Am Ende des Gangs befindet sich eine weinrote Tür. Unten in der Ecke ist Maggie von den Simpsons zu sehen – ein Hinweis darauf, was sich dahinter befindet? Beim Öffnen der Tür erklingt ein Brummen. Große Schränke kommen zum Vorschein, in denen sich viele Kabel verstecken, durch die feinvergitterten Schranktüren sind kleine Lichter zu sehen. Wir befinden uns im Reich der Hochleistungsrechner der FAU.
Hier stehen Emmy und Meggie, zwei FAU-Supercomputer. Emmy ist nach der berühmten Erlanger Mathematikerin Emmy Noether benannt, Meggies Name leitet sich von ihrem Hersteller Megware ab – und nicht wie der Sticker suggeriert von Maggie Simpson.

Solche Hochleistungsrechner sind nicht aufgebaut wie die Computer, die in unseren Haushalten oder an unseren Arbeitsplätzen stehen: „Man muss einen normalen PC hernehmen und eigentlich fast alle Komponenten bis auf die Prozessorkerne und den Speicher wegnehmen. Diese setzt man dicht gepackt in einen Schrank und dann ganz viele dieser Schränke nebeneinander. Das ist ein Hochleistungscomputer“, erklärt Prof. Dr. Gerhard Wellein, Professur für Höchstleistungsrechnen. Viele solcher Schränke stehen im Serverraum des Regionalen Rechenzentrums Erlangen (RRZE). Ihr eher unspektakuläres Aussehen verrät dem Beobachter nicht, was sich Großes in ihnen versteckt – dass in ihrem Innern gerade Eiweiße, Partikelstürme, Strömungsfelder oder Klimaveränderungen simuliert werden.
Das große Wirkstoff-Puzzle

Mithilfe solcher Supercomputer, die der ganzen Universität offenstehen, ist es Wissenschaftlerinnen und Wissenschaftlern der FAU möglich, Herausforderungen anzugehen, die auf einem normalen Rechner nicht lösbar sind, weil dieser zu wenig Leistung hat. Wenn ein Supercomputer des RRZE eine Woche für eine Aufgabe rechnen muss, braucht ein Heim-PC etwa 2000 Wochen dafür – etwas mehr als 38 Jahre.
Einer dieser Herausforderungen hat sich ein Forschungsteam um Dr. Anselm Horn, wissenschaftlicher Mitarbeiter an der Professur für Bioinformatik, gewidmet. Die Forschenden simulieren die Bewegung der Proteine, die im Zusammenhang mit Alzheimer auftreten, am Großrechner. Im Körper eines jeden Menschen lässt sich das Eiweiß Beta-Amyloid finden. Durch Mutationen oder äußere Einflüsse kann dieses Protein jedoch seine Form ändern. Es wird an den Enden klebrig und bildet Stapel, die aufeinanderliegenden Hufeisenmagneten ähneln: sogenannte Fibrillen. Diese Ansammlungen wirken neurotoxisch, können vom Körper nicht mehr abgebaut werden – und sind charakteristisch für die Alzheimer-Krankheit.
Um solche Proteinansammlungen im Laborexperiment untersuchen zu können, brauchen Wissenschaftlerinnen und Wissenschaftler große Mengen an Eiweißen mit identischer Form. Falten sich körpereigene Stoffe um, bilden sich jedoch nicht immer die gleichen Strukturen, was die experimentelle Erforschung erschwert. Außerdem ist es aufwendig, diese Eiweiße im Reagenzglas zu verändern, beispielsweise zu kürzen, und dann deren neues Verhalten zu untersuchen, da sie womöglich verkleben. Eine Computersimulation hat diese Probleme nicht. Dennoch ist sie rechnerisch anspruchsvoll: Die Eiweiße bestehen aus einer Großzahl von Atomen, jedes einzelne muss simuliert werden, ebenso wie seine Bewegung und Wirkung auf die anderen Teilchen. Weil diese Bewegungen immer wieder neu errechnet werden müssen, können nur kleine Zeiträume, also Mikrosekunden, simuliert werden.
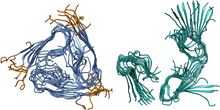
Im Rahmen eines Projektes hat das Team um Dr. Horn eine bestimmte Fibrillenform, die ursprünglich im Gehirn eines Patienten gefunden wurde, betrachtet: das U-förmige Beta-Amyloid, das drei Stapel bildet, die sich so zusammenlagern, dass ein Kanal in der Mitte entsteht. Das Team hat in der Simulation die Enden abgeschnitten und untersucht, ob das Protein noch immer stabil ist. Das Ergebnis: ist es nicht. Wenn der Eiweißbaustein Beta-Amyloid in der Fibrille gekürzt wird, klappt der Stapel auf und geht kaputt. „Die Ergebnisse sind ein Beitrag zum großen Puzzle der Alzheimer-Wirkstoffforschung“, sagt Dr. Horn. Denkbar wäre nun, einen Stoff zu entwickeln, der die Bildung solcher Fibrillen-Strukturen verhindert oder sie so verändert, dass sie zerfallen. Je mehr verschiedene Betas-Amyloid-Strukturen bekannt sind, desto präziser kann ein späterer Wirkstoff funktionieren. Die Ergebnisse bilden folglich eine wichtige Grundlage für Wirkstoffdesigns: „Im besten Fall sollte ein Wirkstoff wie ein Generalschlüssel wirken und für möglichst viele schädliche Eiweißverklumpungen gleichzeitig funktionieren “, erläutert Dr. Horn.
Zwar können solche Computersimulationen Experimente nicht ganz ersetzen, sie können aber die Forschung erleichtern. Besonders, wenn es so fair zugeht wie an der FAU. Während Dr. Horn früher teilweise am Wochenende nachts um zwei Uhr Rechenzeit spontan genutzt hat, wenn sie gerade frei geworden ist, kann er heute dank der guten EDV-Infrastruktur des RRZE die Rechnungen automatisch starten lassen und dadurch durchschlafen. Denn ein einzelnes Projekt rechnet nur jeweils 24 Stunden am Stück, dann wird es im Hintergrund gespeichert und andere Forschende bekommen Rechenzeit. Die Warteschlange ist noch dazu intelligent und verhindert eine „Monopolisierung“ des Rechners durch eine Arbeitsgruppe: „Wer mehr rechnet, rutscht nach hinten. Wer weniger rechnet, nach vorne“, erklärt Prof. Wellein. Auf diese Art und Weise lassen sich die RRZE-Ressourcen ideal nutzen. Im Jahr 2018 griffen mehr als 70 Projektgruppen auf diese Ressourcen zu – dazu gehören Wissenschaftlerinnen und Wissenschaftler aller Fakultäten sowie Studierende, die für ihr Studium oder ihre Abschlussarbeiten rechnen. Die Rechner sind also stets gut ausgelastet.
Ein regionales Modell

Viel rechnen muss auch das Team um Prof. Dr. Thomas Mölg, Institut für Geographie, das sich mit Klimasimulationen beschäftigt: Wie wird sich das Klima in Zukunft verändern? Und wie stark hat der Mensch diesen Wandel bisher beeinflusst? Solche Einschätzungen sind mithilfe von Klimamodellen möglich. Auf globaler Ebene liegen bereits viele solcher Modelle vor. Seit den 1970er-Jahren sind die weltweiten klimatischen Veränderungen eindeutig menschlichen Ursprungs, ob sich diese aber lokal verstärkt oder abgeschwächt äußern, ist weitestgehend unbekannt. Das wollen die Forschenden ändern und ein Modell entwickeln, das die globalen Signale auf eine regionale Ebene bringt. Als Beispiel dienen die Hochgebirge Ostafrikas.
Globale Klimamodelle bestehen aus einzelnen Punkten, für die der klimatische Zustand – also zum Beispiel Meeresströmungen, Temperaturen, Winde – errechnet wird. Der Abstand zwischen den Rechenpunkten beträgt im besten Fall 30 bis 40 Kilometer. Für regionale Betrachtungen sind diese zu groß, die Punkte müssen näher zusammenrücken. „Man kann sich das wie bei einem Foto vorstellen: Will man ein Detail eines Fotos näher betrachten und zoomen, muss die Auflösung groß genug sein“, erklärt Mölg. Um die regionalen Modelle zu erstellen, nutzen die Wissenschaftlerinnen und Wissenschaftler globale als Grundlage, die sie mit Daten aus eigenen Messungen vor Ort über einen Zeitraum von 20 Jahren ergänzen. Dann kommen die Supercomputer ins Spiel: Prof. Mölg will alle 500 Meter das Klima simulieren, dementsprechend viel Rechenleistung wird gebraucht. Solche Simulationen wären theoretisch zwar auf normalen Computern möglich, praktisch jedoch kaum zu realisieren – schon allein wegen der jahrelangen Rechenzeit. Die zwei Supercomputer der FAU sind jedoch gewappnet, sie gehören regelmäßig zu den 500 schnellsten weltweit. Und der nächste Supercomputer ist bereits in Planung: ein Rechner im Wert von 4,5 Millionen Euro soll angeschafft werden. Prof. Mölg lobt die High-Performance-Computing-Ressourcen: „Das war für mich auch ein Grund, an die FAU zu kommen“, sagt er.
Die klimatischen Achillesfersen

Je näher man jedoch hereinzoomt, umso störender werden dann auch andere Einflüsse. Eine Herausforderung ist also, ob man überhaupt noch einzelne Einflüsse unterscheiden und diese eindeutig zuordnen kann. Die Wissenschaftlerinnen und Wissenschaftler müssen folglich lokale Besonderheiten herausfiltern. Eine besondere Rolle spielen hierbei die „Achillesfersen“, die Mechanismen, die globale Signale in eine Region übertragen und daher besondere Bedeutung für die menschliche Klimabeeinflussung besitzen, wie beispielsweise der Feuchtigkeitstransport in den unteren 1000 Metern des Indischen Ozeans für das Beispiel Ostafrika.
Im Idealfall ist die Methodik der FAU-Forschenden auf andere Gebiete übertragbar. „Das Suchen der ‚Achillesfersen‘ ist auch für den Raum Erlangen-Nürnberg denkbar“, erklärt Prof. Mölg. Doch bis dahin wird noch einige Rechenzeit vergehen. Und wenn das Licht ausgeht, die weinrote Tür zufällt und das Brummen verstummt, scheint es wieder schier unmöglich, dass dahinter vielleicht gerade digitale Stürme toben oder flüssige Metalle wabern – und dass die Forschung in diesem Raum niemals schläft.
Ich sehe eine Farbe, die du nicht siehst …?!

Ist die Tür eher lila oder eher rot? Hat sie die Farbe einer noch nicht ganz reifen Aubergine oder die eines guten Rotweins? Und würde beispielsweise die Farbe einer Aubergine auch wieder gleich benannt werden? Das haben wir uns während der Textredaktion gefragt – und: Wie nehmen Menschen Farben überhaupt wahr? Prof. Dr. Jan Kremers, Lehrstuhl für Augenheilkunde, hat es uns erklärt: Wir erkennen Farben mithilfe der Zapfen in der Netzhaut, die entweder auf kurze, mittlere oder lange Wellenlängen reagieren. „Wir sehen die Farbe Rot, wenn die Zapfen, die auf langwelliges Licht ansprechen, stärker erregt werden als die anderen“, erklärt er. Bei Blautönen werden hingegen die Zapfen, die für kurze Wellenlängen empfindlich sind, angesprochen. Die Zapfen selbst unterscheiden dabei keine Farben; der Farbeindruck wird errechnet, indem die Aktivität der unterschiedlichen Zapfen verglichen wird. Wie wir eine Farbe individuell benennen, ist zudem kulturell bedingt: Wir erlernen die unterschiedlichen Farbbezeichnungen in der Kindheit. Doch ob das Weinrot der einen auch das Weinrot der anderen ist, kann nicht genau gesagt werden. „Die Benennung und die subjektive Wahrnehmung sind ein ganz anderes Thema“, sagt Prof. Kremers.
FAU-Forschungsmagazin friedrich
Dies ist ein zusätzlicher Online-Artikel zu unserem Forschungsmagazin friedrich. Die aktuelle Ausgabe nimmt Sie mit auf eine Entdeckungsreise ins „Verborgene“: Sie schaut auf für unser Auge unsichtbare, oftmals von uns unbemerkte und vor uns versteckte Dinge. Sie wirft aber auch einen Blick dorthin, wo wir gar nicht hinsehen wollen: auf Tabus.
Ein Print-Exemplar können Sie sich direkt an einer der vielen Auslagen der FAU mitnehmen oder kostenfrei bestellen.

